前言
“课题做不下去了,要不测个序吧!”
作为一个分子生物学实验室出来的学渣,在学生生涯的最后几年,经常能听到这句话。
随着RNA测序的价格愈发走低,在缺乏明确目标的情况下,RNA测序已逐渐成为分子生物学课题中,筛选后续研究方向最为省时省力、经济实惠的手段。研究者往往使用RNA测序来推进项目的进展,然而,许多新入行的研究者对RNA测序的原理知之甚少,应用也仅仅停留在获取基因表达量的水平上,对于二代测序技术来说,这无异于莫大的浪费。
这个系列的文章,就是想跟大家简单聊聊RNA测序的数据如何生成,如何分析,以及除了作为表达芯片的替代品外,还能为我们带来哪些有用的信息。第一篇就先简要介绍一下RNA测序大致的流程,以及RNA测序(与价格相关的)分类。
一、二代测序的原理
虽然二代测序仪的厂商曾经有很多家,但核心的原理大同小异,基本都是利用高通量测序获取样品中RNA的序列信息,测到次数越多的序列,表达量也越高。下文将以市场占有率最高的Illumina测序平台为例,简单介绍我们提取的RNA是如何变成测序文库(library),再变成硬盘中的数据的。先上图:
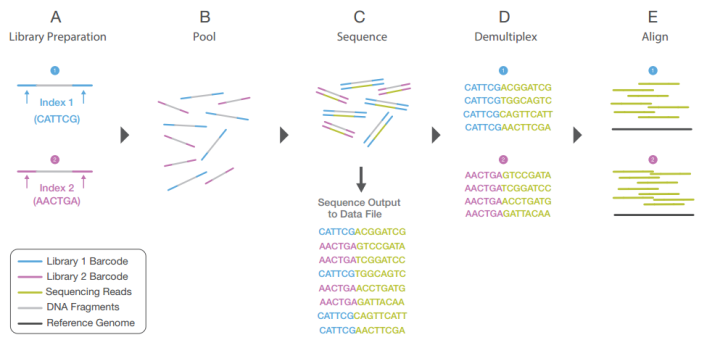
上图简单的描述了二代测序的大体流程,即“文库构建→混合上机→获取序列→数据分割→分析比对”。我们这部分主要讲一下前三个步骤,涉及到数据分析的内容,我们留到第二篇文章再讨论。
二代测序需要制备能够被测序仪直接分析的带有特定序列并在一定长度范围的测序文库,因此我们需要结合RNA富集、片段化、反转录、加接头、PCR扩增、长度选择等方法,获取带有接头序列(用于桥式PCR扩增)、Barcode序列(用于区分不同样品)以及插入的待测cDNA片段的序列。具体步骤可以去这个网址戳说明书(https://www.illumina.com.cn/products/by-type/sequencing-kits/library-prep-kits/truseq-rna-v2.html),常规的测序文库长这样(不同建库试剂的文库结构存在差异):

文库构建完成后,就可以把不同样品的文库混合,并上机测序。Illumia测序原理如下图所示,简单概括就是将文库结合到测序芯片上,并通过PCR将单一序列扩增成簇以提高信号强度(具体步骤请搜索“桥式PCR”),然后测序时收集每一簇的荧光信号,并转换为相应的碱基,从而获取测序数据。
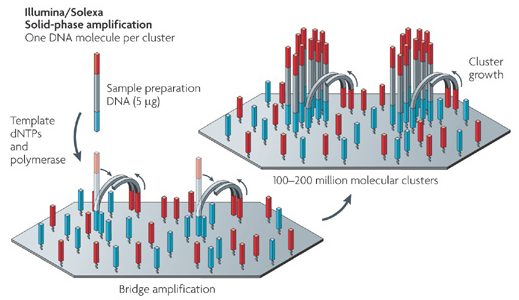
图片来源:http://www.nature.com/nrg/journal/v11/n1/full/nrg2626.html
二、RNA测序有哪几类
常规的RNA测序分三大类:转录组测序(我们平时最多提到的RNA-seq)、小RNA测序、以及近两年比较火热的环状RNA测序。这三大类测序的目标分子差别很大,因此一般也不会出现混淆。
其中转录组测序主要针对线性的mRNA以及lncRNA(长非编码RNA),基本的流程就是我们刚才介绍的;小RNA测序主要针对18~40个nt的小分子RNA(比如miRNA、snoRNA等),需要在构建文库之前通过片段选择富集小分子RNA;而环状RNA测序则需要先使用RNase R降解掉线性RNA分子(之前笔误写成降解DNA-RNA杂合链的RNase H了,感谢 @春宇 指出,顺便有些公司去除核糖体RNA用的就是RNase H),再进行建库测序。此外,根据研究目标的不同,还有诸如RIP-seq、CLIP-seq等不同应用,具体文库的构建方式,需要针对不同的应用场景进行优化。
三、同样是转录组测序,为什么我做的比别人贵那么多?
一个样品的RNA测序几千块钱的花销对于一个课题来说也是一笔不小的支出,而有时我们会发现有的公司的报价连其它公司的一半都不到,这是为什么呢?
影响RNA测序成本差异的因素主要有下面四个:
1. 数据量不同
这个比较容易理解,毕竟测的数据量越大,价格就越高。一般国内的测序公司计算数据量时使用两套标准:碱基数(base,通常以G为单位)和序列数(reads,通常以M为单位)。换算也很简单,序列数×序列读长=碱基数(举个例子,我做了20M reads的转录组测序,序列读长是双端150bp,那么碱基数就约等于2×150×20M=6G)。
顺便说一句,对于人、大鼠、小鼠这些成熟的物种,只研究mRNA表达量,大概6G碱基就足够了,研究lncRNA表达建议12~15G。对于小RNA测序,5~10M reads基本能够满足要求。至于环状RNA,祝你好运…
2. 测序平台不同
这个也好理解,不同测序仪的测序成本是存在差别的,比如用Illumina HiSeq X Ten的成本差不多只要Illumina HiSeq 3000/4000平台的一半,而更早的HiSeq 2000成本更是惊人。此外MiSeq平台由于读长更长,因此同样reads数的成本也大幅高于HiSeq平台。最后BGISEQ-500平台最近价格肯定有优势,但我没用过,有用过的可以聊聊感受。
3. 文库类型不同
这一点可能是对价格影响最大的因素。由于很多细胞和组织中,70%以上的RNA是核糖体RNA,因此在文库构建阶段,有必要去除核糖体RNA带来的干扰。一般来说,有两种方式:① oligo(dT)捕获技术:利用mRNA通常都带有poly(A)尾巴的特性(不知道poly(A)是干嘛的请自觉找吴思涵老师补课),将mRNA(和部分lncRNA)与总RNA分离;② 核糖体去除技术:使用针对核糖体RNA设计的探针,特异性去除总RNA中的核糖体序列。
通常来说,方法二的成本显著高于方法一(加上方法二由于测到的转录本更多,因此通常会测更多数据,价格翻倍都是很正常的),相应的,核糖体去除技术能够带来两点显著的优势:① 转录本覆盖均一性好:由于oligo(dT)捕获技术容易导致RNA分子断裂,因此oligo(dT)捕获得到的转录本通常在3’端的测序深度显著高于5’端,对定量分析存在一定的干扰;② 能够测到大量无poly(A)结构的RNA:由于一大部分lncRNA不含poly(A)结构,因此只有通过核糖体去除技术才能进行测序,此外,环状RNA也可以使用核糖体去除技术进行测序(不过定量就别想了,不准)。
还有一个影响价格的因素是“链特异性文库”的构建,由于RNA可能由DNA模板的两条链转录,但传统的建库方式无法区分测到的RNA序列源自哪条链,因而丢失了很多数据分析的过程中重要的信息,而通过链特异性文库构建的方法也增加了成本。
所以,下次销售们报出价格的时候,你大概能够分辨到底值不值了吧?
Q&A
Q1. 既然是看表达量,为什么我不用表达谱芯片(RNA microarray)呢?
简单来说,相对于表达谱芯片,RNA测序有下面三方面优势:
\1. 测序的定量更准确,与表达量检测的金标准——定量PCR——一致性更高,可以戳这篇文章看细节(A comprehensive assessment of RNA-seq accuracy, reproducibility and information content by the Sequencing Quality Control Consortium.);
\2. 表达谱芯片只能对已知的转录本进行定量,对未知的转录本或难以设计探针的转录本无法检测(更何况有时候遇到某些芯片公司坑爹的探针,真的欲哭无泪);
\3. 除计算表达量外,RNA测序还能做很多其它的分析,也许某天你重新翻翻以前的数据,还能再发一篇小文章。
Q2. 所以测序到底还能多做什么?
说几个常见的项目:① 可变剪接 ② 新转录本鉴定 ③ 融合基因分析 ④ SNP检测
其他项目,可以问问测序公司的销售他们能做什么,他们会很乐意告诉你的。
